Term-2; For 3rd/4th year undergraduates (and Master course Grads)
Text: GENE CLONING BY
T.A. BROWN from Chapman and Hall (1999)
Part I.
The Basic Principles
Part II.
The Applications
Part III.
In Our real World
Part IV.
Our Genetic Future
Aims: To learn laboratory
techniques
To discuss
ethical matters
Practice; Optional
- 약 1세기 전 멘델은 유전의 기초 원리를 발견
- 유전적 특성; 유전자
- 1900 유전법칙의 재발견; 유전학의 탄생
1.1 유전학의 발달 초기
- 서튼, 1903년, 염색체의 존재를 주장

- 모간, 1910년, 실험적으로 존재의 확인
- 모간, 1922년, 유전자 지도 작성기술 개발 ; 2천여
개의 초파리
유전자 지도 작성
- 에이버리, 맥리오드, 맥카티, 1944년
- 허쉬, 체이스, 1952년; DNA가 유전물질임을 입증
(Not Protein)
- 델뷰릭, 챠가프, 크릭, 모노드; 유전학 발전의
도약기에 기여
- 1952-66 (14년간); DNA 구조 규명, 유전 암호의
해독, 전사와 해독의 과정이 밝혀짐.
1.2 Gene cloning의 진보
- Revolution in experimental biology for genetic
research, 1971-73년
=> Recombinant
DNA technology/ Genetic engineering/gene cloning
- 이후 30여년간 놀라운 발전을 가속화 하고 있음.
1.3 Gene Cloning 이란?
- 그림 1. 1, basic steps
1.4 Gene cloning은 특별한 도구와
기술이 요구된다
1.4.1 운반기구
- 유전자를 숙주 세포로 운반해 주는 기구; 벡터 (vector)
=> 숙주로의 이동이 가능하여야 하고
=> 자신과
같은 복제품을 만들 수 있어야 한다.
- Plasmids
- Virus chromosome (bacteriophage)
=> See
CH2 for basic features
1.4.2 DNA를 다루는 기술
- 실험적으로 잘다룰줄 알아야만 한다
- Manipulative skills (표 1. 1)
=> 고순도의 DNA 정제
기술 (3장)
=> 제한효소, 봉합효소
(DNA의 재단)
(4장)
=> 숙주로의 운반 (형질전환에 의한 방법) (5장)
1.4.3 Cloning Vector의 다양함
- 자연산 plasmid/viruses을 변형하여 사용함
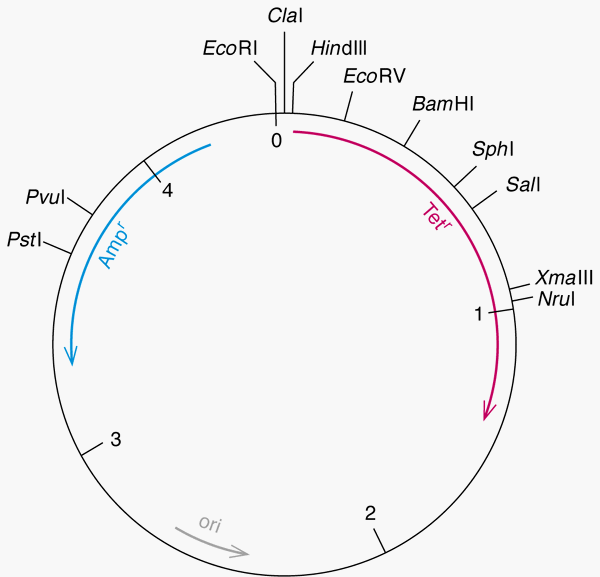
- 용도에 맞는 것을 선택해야함 (6, 7장)
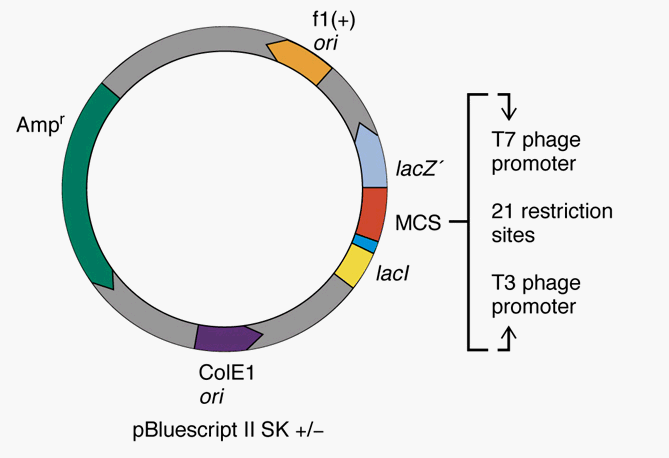
현재 가장 많이 사용되는 플라스미드 벡터인 Bluescript
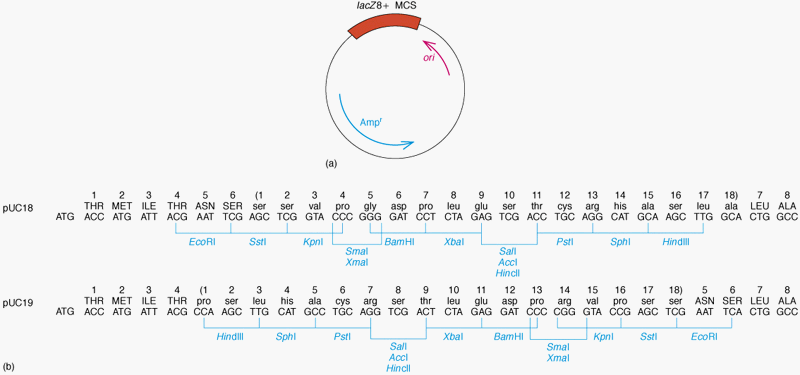
MCS (Multi Clining Sites)
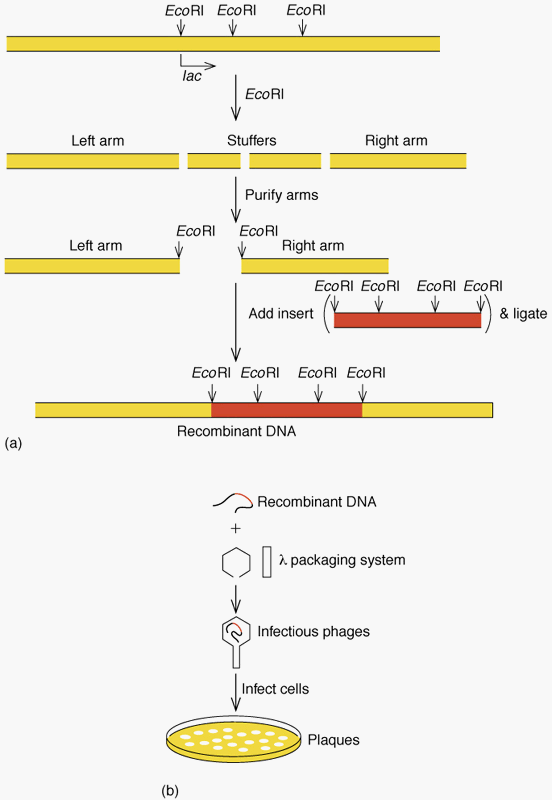
Phage lambda vector를 이용한 유전자 은행의 제조 과정의 일부
1.5 Gene Cloning은 왜 중요한가
- 순수한 유전자를 분리할 수 있음
- 그림 1. 2
- 부가 설명
- 그림 1. 3; 대장균의 유전자 도서관 작성 및
선별
- 8장에서는 다양한 기법을 소개함
- 한 유전자의 분리는 가히 무한정의 연구를 가능케함
=>구조, 발현 등 (9,
10장)
1.6 Polymerase Chain Reaction (PCR)의
중요성
-PCR 기법의 개발, 1980 년대 말
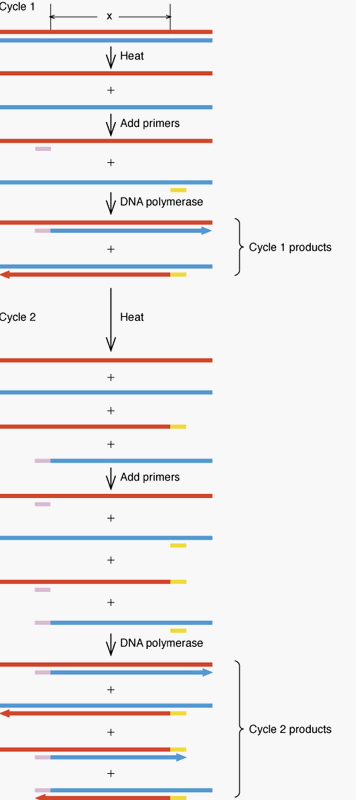
=> can amplify DNA
and genes. (11장)
=> 그림 1. 4
1.7 과학적연구와 생명공학에 재조합
DNA 기술이 가져다 준 충격
- 생물학의 연구에는 광범위하게 적용되고 있다
- 산업에도 응용
; 유용 단백질의 생물공학적 양산이 가능해짐
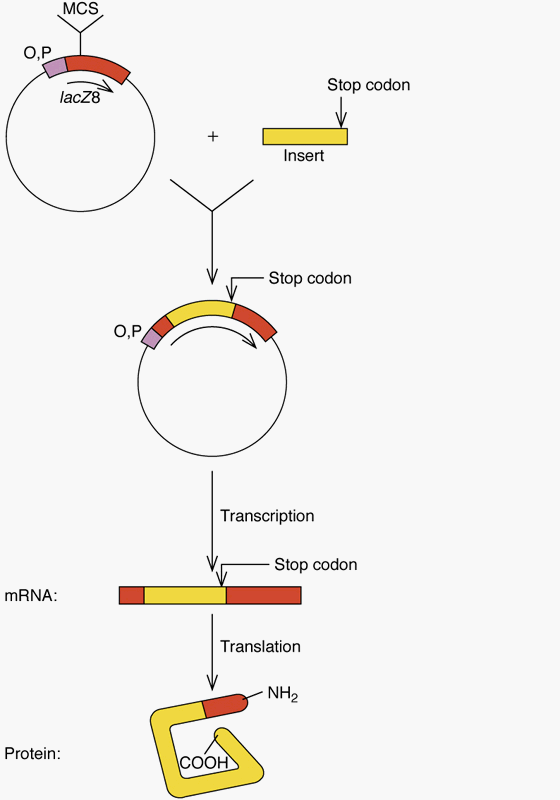
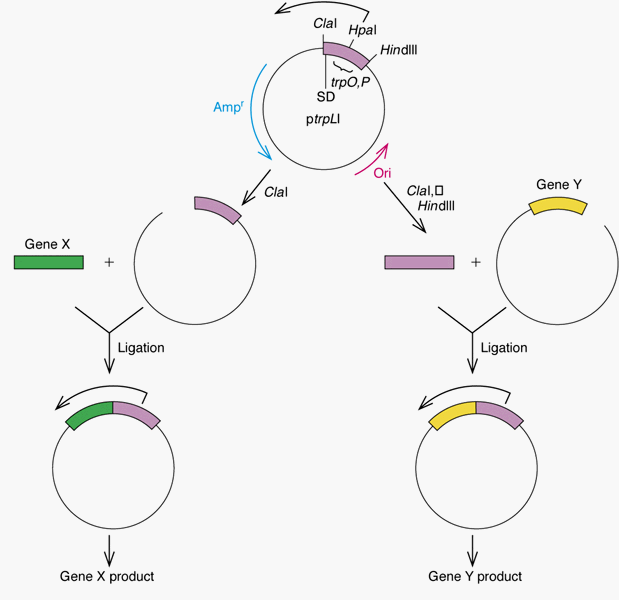
=> 동물 단백질의 대장균내
생산 (그림 1. 5)
=> 의약품, 백신, 호르몬 생산,
태아의 유전학적 검사, 암의 치료, 유전병의 치료,
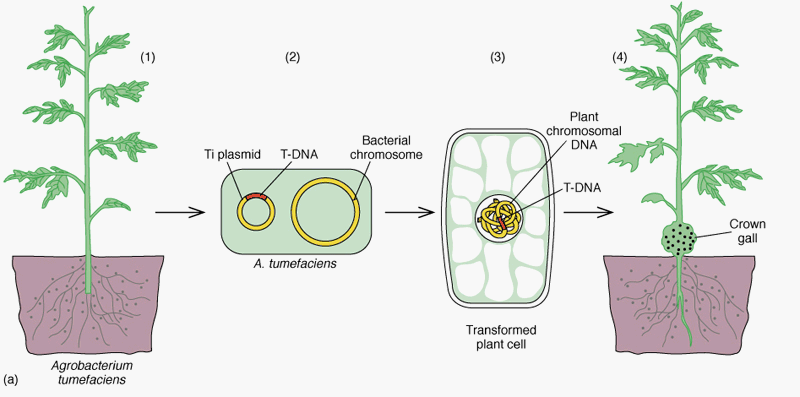
=> 농업적 유용 작물의 개발 (12-14장)

=> Crown gall in tobacco plant
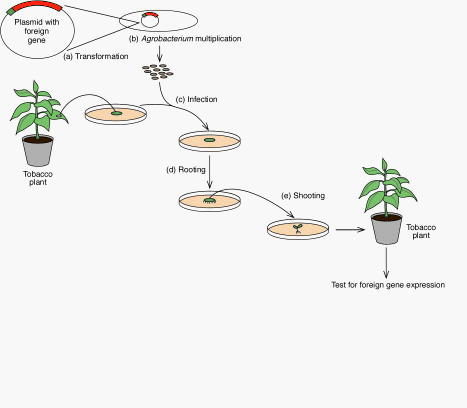
식물로의 인위적 유전자 도입 과정
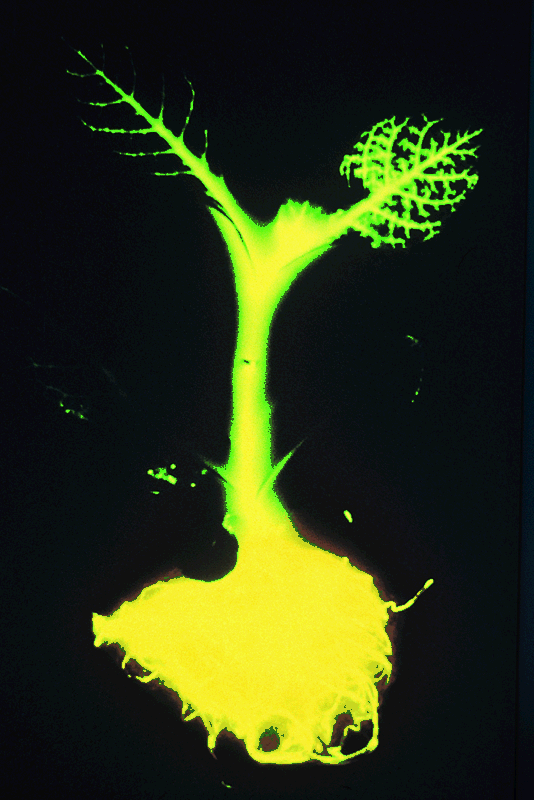
곤충(반디)의 유전자로 형질전환된 담배의 유식물체
제 2 장
핵산의 운반 도구: 플라스미드와 박테리오파아지
** Gene cloning을 위한 DNA가 가져야 할 특징;
- 세포내에서 자가 복제; 많은 재조합 DNA 복제품
- 다음 세대로의 유전성
- 작은 것; 10 kb 미만; 클수록 정제과정 중 분해가
쉽고 조작이 어렵다.
- 전통적으로 2가지가 운반기구가 사용됨; plasmids, bacteriophage-chromosomes
2.1. PLASMIDS; independent genetic
elements
2.1.1. Plasmids의 기본적 특징
- 원형 (circular)의 DNA
- 여러개의 유전자를 운반할 수도 있다. (그림 2.
1)
=> 이들 유전자는 숙주
박테리아의 유전자적 특징을 나타낸다.
=> 항생제에 대한 내성 (antibiotic resistance)을 나타냄 (항생제)
=> Selectable marker (그림 2. 2)
- Origin of replication; 독자적인 복제에 필요한
DNA 염기서열이 있어야함.
(그림 2. 3a, b)
=> episomes; integrative
plasmids
2.1.2. 크기와 복제수
- 매유 중요한 요소임 (크기는 다양; 1kb - 250
kb)
(표 2. 1)
- Gene cloning에 적절한 크기는 매우 제한적
=> 3-5 kb
- Copy number; 대략 50 copy, (다다익선)
2.1.3. Conjugation and compatability
- 접합성; sexual conjugation (그림 2. 4)
=> tra 유전자에 의해
조절됨(접합성 plasmid에만 있음)
=> 비접합성의 plasmid 또한 접합성 plasmid가 있을 때 동시에 전이가 가능함
- 여러 종류의 plasmid가 하나의 대장균 세포에
공존할 수 있다.
(현재까지 알려진 바로는 최대 7종류)
=> Plasmid의 상호 공존성을
compatibility라함.
=> compatibility의
이유는 잘 알지 못함.
2.1.4. Plasmid의 생물학적 분류
- 기준; 유전자의 특성
- 약 5 가지로 분류가 가능하다.
1) ‘F’ plasmids(Fertility); gene만
있음
2) ‘R’ plasmids(Resistance); 항생제 내성
유전자
=> 클로람페니콜, 암피씰린, 테트라싸이클린
=> 임상미생물학에
매우 중요
=> 전파를 방지해야함
3) Col plasmids; code for colicins
=> colicin은
다른 박테리아를 죽이는 단백질
=> ColE1
대장균
4) Degradative plasmids; 톨루엔이나 살리씰산
등 희귀물질을 대사할 수 있는
=> TOL of P.
putida
5) Virulence plasmids; pathogenicity
=> Ti-plasmids
of A. tumefaciens
(쌍자엽 식물에 종양을 유발함)
2.1.5. 박테리아 이외에 존재하는 plasmids
- 효모에 있는 2um circle
- 현재까지 다른 동물에는 없다 (?).
2.2. BACTERIOPHAGES
2.2.1. 박테리오파이지의 기본적인 특징
-파이지=바이러스
-숙주 특이적인 박테리아에 감염
-Simple structure; DNA or RNA + capsid
(그림 2. 5)
-감염의 과정; 3단계 (그림 2. 6)
=> lytic cycle (용균성
회로)
2.2.2. Lysogenic phages (용원성 회로)
-숙주내 파이지 DNA를 보유한 상태로, 숙주 세포의
분열과 동행한다.
-파아지 DNA는 박테리아 제놈내로 삽입
=> episomal insertion
(그림 2. 3b)
=>
called prophage (integrated form of DNA); 잠복 상태
=>
결국 prophage는 숙주인 lysogen의 제놈으로부터 방출되는
용균과정으로 되돌아가 숙주를 파괴한다. (예; 람다 파아지)
=>그림 2. 7
-M13 phage의 경우는 다른 형태의 lysogeny를 경유한다.
=> 숙주내에서 복제와
조립이 되고나서 방출을 계속.
=> DNA는 숙주 게놈으로
삽입이 일어나지 않음.
=> 그러나 cell lysis는
일어나지 않음.
=>
감염된 숙주는 계속적으로 생장, 분열. (그림 2. 8)
-현재까지 람다와 M13 만이 유용한 벡터로 활용됨.
(a) Lambda DNA의 유전자 구성
-a typical head-to-tail phage (그림 2. 5a)
-감염의 경로; (그림 2. 7)
-49kb (유전자 지도, 그림 2. 9)
=> genes are clustered
(유전자들이 무더기로 존재)
=>
유전자의 발현 조절 및 람다-based cloning vector의 구성에 중요
(6장 참고)
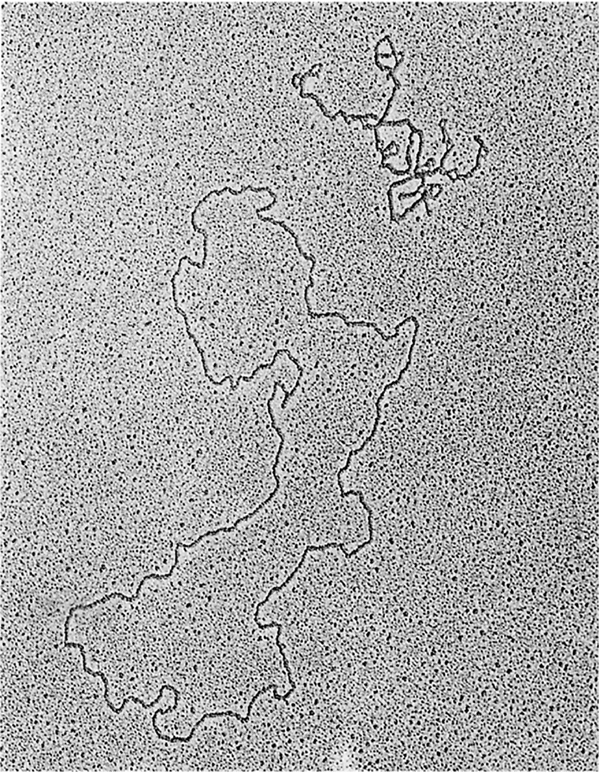
(b) 람다 DNA의 linear and circular forms
-linear structure; two free ends
=> DNA 분자의 5’말단에
phosphate group제거
2) Polynucleotide kinase; from T4 phage
=> Alkaline phosphatase와
반대의 기능
3) Terminal deoxynuleotidyl transferase; from
calf thymus
=> DNA 분자의 3’말단에
한개 이상의 DNA 첨가
4.1.5. Topoisomerases
cccDNA (plasmid) 의 형태 변형; by supercoil의
도입/제거
=> 유전자 조작에서 유용성은
없음.
4.2. Enzymes for cutting DNA;
제한효소
- 절단하고자 하는 DNA의 정확한 위치 잡기/자르기
=> vector의 한곳 자르기; 외래 DNA의
삽입 가능
=> 외래 삽입 DNA의 정확한 자르기
(그림 4. 7)
- 특정한 형태의 nuclease를 요함
- 원하는 DNA를 cloning하기 위하여;
1) DNA의 절단이 요구됨; genomic
DNA (--80 kb)
2) 절단시 vector로 삽입/운반
가능한 크기로
=>
vector마다 삽입 가능한 크기가 있음.
=> 예; M13의 경우 3kb이상 고순도의 정제된 제한효소가 필요함;
유전공학발전의 전기가 됨 (1978 노벨상; Arber, Smith, Nathans)
4.2.1. The discovery and function of REs
- 1950s; 파아지의 숙주 제한성 (host-controlled restriction)
=> 일부의 박테리아가
파지의 감염에 대하여 면역성을 띰.
=> 제한효소가
파지 감염 후 파지 DNA의 복제 이전에 파괴 (그림 4. 8a)
=> 숙주
DNA는 메틸화해 제한효소로 부터 보호된다. (그림 4. 8b)
=> 대부분의
박테리아에 있는 면역기능.
=>1200여 종류가 밝혀짐.
- Three types; I, II & III
1) I & III; complex and limited for genetic
research
2) II; important for gene cloning
4.4.2. Type II RE cut DNA at specific..-
- 각각은 특정 인식부위가 있음; mostly hexanucleotide
some at 4, 5, or 8 nucleotide
seq.
ex) PvuI (from Proteus
vulgaris) cut only CGATCG
PvuII;
cut CAGCTG
Sau3AI
(From Staphylococcus aureus strain 3A)
=> cut at GATC
AluI
(from Arthrobacter luteus); cut at AGCT
- 관련부위를 자르기도함
예) HinfI (from Haemophilus..
..)
=> cut at GANTC
표 4.1; 흔히 사용되는 제한효소
목록
4.2.3. Blunts ends and...
- blunt or flush end; PvuI and AluI; blunt-end cutters
- sticky or cohesive end; ssDNA staggered ends (그림
4. 9a, b)
어떤것은 동일한 sticky end를 만듬
=> 그림 4. 9c, 이들의 결함은
모두 가능함.
4.2.4. The frequency of .......
- 특정한 제한효소에 대한 절단횟수; 수학적
=> at random and GC=50%일
경우
=> GATC; 44=256에 1회
=> GGATCC; 46=4096에 1회
=> 실제는 그렇지 않음
예;
lambda DNA(49kb); hexa가 12회 이어야함
=> BglII=6, BamHI=5, SalI=2
- 제한효소의 부위는 불규칙적임;
=> 자른 후의 분절 크기가
다양해짐 (그림 4. 10)
- 실험
4.2.5. Performing a .....
-test-tube, micropipet, tips,
-H2O,
-REs; 1 g/unit
-buffer; 10배 용액을 구입
=> pH 7.4
=> different ionic strength,
NaCl
=> Mg++ ions for type II
=> reducing agents; DTT (효소의
안정화, 불활성화 방지)
=> 부정확시 RE의 활성저하 및 불균일 절단
(예) 표 4. 2
-DNA
-37도 C 배양기 (30-42-65C); 1-4h
-예; 20 l reaction (그림 4. 11)
-처리 후 제한 효소 활성 제거; 70C, EDTA, phenol
4.2.6. Analysing the result of RE cleavage
-Number and size
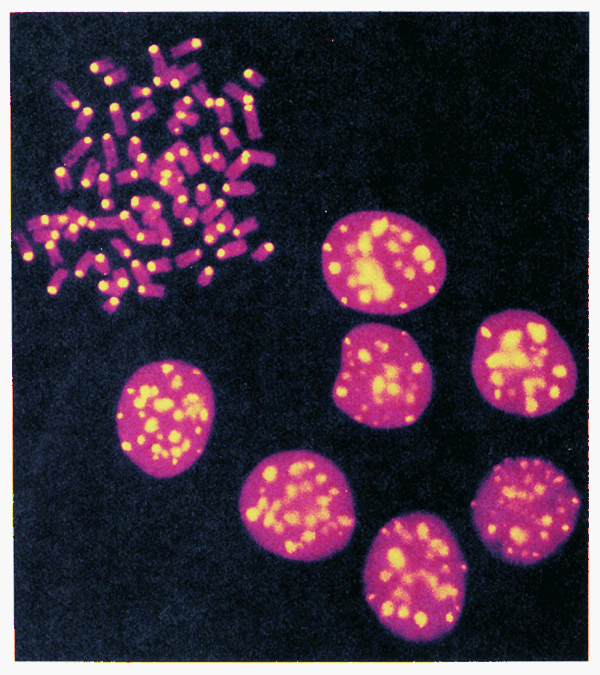
-Gel electrophoresis; 1970년대 초 개발
(1) 전기영동에 의한 분자의 분리
- negative charged DNA
=> 전기장 내에서 양극
쪽으로 이동 (그림 4. 12)
=> 이동의 속도; 분자의
모양과 charge to mass
=> 이 경우 charge to mass ratio가 유사함
=> 정상적인 전기 영동에서는 분리 불가
- 그러나 분자의 크기가 중요한 인자가 된다
=> agarose/polyacrylamide;
pore로 구성됨
=> 작은 분자가 빨리 이동함
=> agarose; 비교적
큰 pore>작은것 분리에 한계
=> 1-30kb
=> polyacrylamide;
very pore
=> can separate 1 to 300 bp ragne
=> even single n.t. difference
(2) Visualizing DNA.....
1) Staining; EtBr and UV (그림
4.13); limit its sensivity
=> 25 ng
이상에서만 유효
2) Autoradiography of ...
- 전기영동전에
DNA의 방사선 표지(by incorporation)
=> X-ray photo film에 노출 (그림 4. 14)
- 32P;
=> nick translation (DNA pol. I) (그림 4.15b)
=>any DNA can be labelled
=> end-filling (by Klenow frag.)
=> only for sticky-end fill-in (상보가닥)
(그림 4.15c)
- 2 ng의 DNA도
검색 가능함.
4.2.7. DNA 분자의 크기 판별
Use standard restriction digest; Lambda DNA
Hind III
=> 8개 의 분절 형성 (그림
4. 16)
4.2.8. Mapping the positions of different restriction
sites on a DNA molecule
- Restriction analysis
- Restriction map and mapping
- Double digestions
- Partial digestion
(그림 4. 17/4. 18)
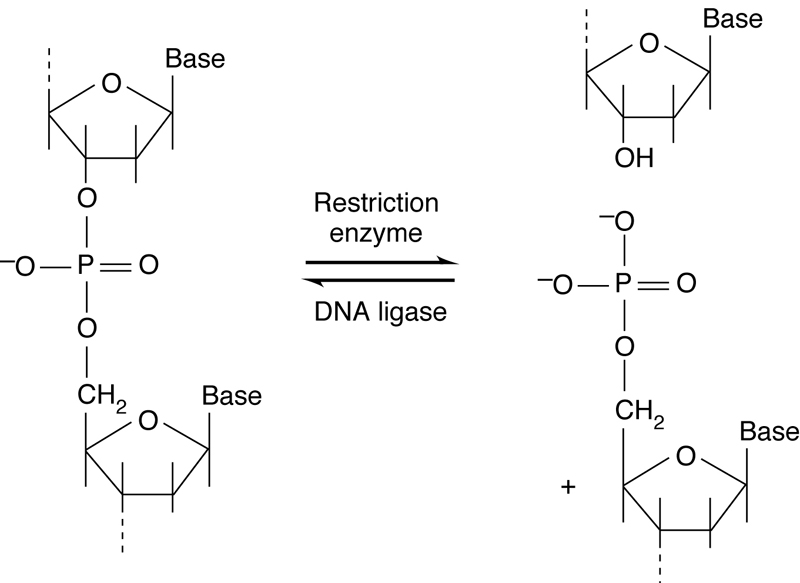
4.3. Ligation-Joining DNA ...
- Final step: joining together (그림 4.
19)
4.3.1. The mode of action..
- Ligase는 모든 생물에 있다
- 실험용은 T4 phage에 감염된 대장균으로 부터 분리
=> phosphodiester bond의
분절화를 봉합
(Nucleotide가
결손되는 Nick와는 구별됨)
- 자연 발생적으로 일어나는 이러한 불완전함은 ligase에
의해 수리되므로 그 역할이
중요하다. (그림 4. 20)
4.3.2. Sticky ends increase the ...
- Blunt ends는 분자간에 catch hold가 없으므로 효율이
낮음 ==>>>
require high conc. of DNA (그림 4. 20)
- 결정요인은 알맞는 비율의 vector/DNA와 온도, 시간
4.3.3. Putting sticky ends on to..
- Vector가 sticky이며 DNA분절이 blunt-ended일 때 3가지의
방법이 있음.
(a) Linkers
- short pieces of dsDNA of
known n.t. seq.
=> 인공합성된것임;
제한효소의 부위를 가짐.
=> 이를
붙이는 것은 비교적 용이
=> 여러개의
링커를 붙일 수도 있음
=> 이후
제한효소의 처리로 sticky-end노출
(그림 4. 21a,b)
(b) adapors
- Linker의 제한효소 부위가
DNA에 존재시 사용 (그림 4. 22)
- short synthetic oligonucleotides
=> 이미
sticky-end를 가짐 (그림 4. 23)
- Require phosphate group
on 5'-OH by polynucleotide kinase
(c) Producing sticky ends by homopolymer tailing
- Using terminal deoxynucleotidyl
transferase to 3'-OH
- and Klenow polymerase
(그림 4. 26)
제 5 장
Introduction of DNA into living
cells
-Cloning; means in bacteria
-두 가지의 목적을 위한것;
1) 대량의 재조합 DNA의 생산 (그림 5.1); 수 천/수
백만배로 증폭 가능함.
=>유전자 구조나 발현의 분자생물학연구의
시료로.
2) 원하는 것(재조합 DNA)의 순수 정제;
=> ligates는 다음을 포함;
(그림 5.2a)
-
ligated vector molecules ; 큰 문제는 아님
-
ligated DNA frags.; 큰 문제는 아님
-
Self-ligated vector; 선별이 요구됨 (그림 5.2b)
-
wrong recombo. DNA; 선별이 요구됨 (그림 5.2b)
=> 대장균은 한 종류의 DNA만을
수용
=> 어떻게 이들을 분별할 것인가?
selection
5.1. Transformation-The uptake
of DNA by
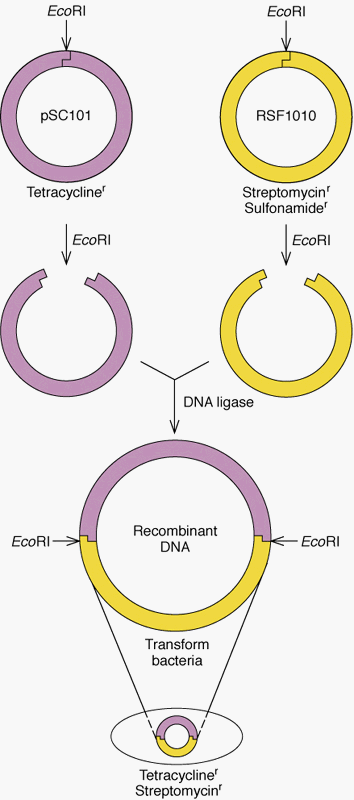
-Plasmid; antibiotic resistant
=> ex) pBR322; ampR, tetR
by beta-lactamase and detoxification
=> 형질전환된 대장균은 두 항생제에 대해
저항성
-Transformation; uptake of any DNA molecule
by any type of cell
=> 의미가 확대되어 동/식물 및 곰팡에에
이르기까지 모든 생물이 새로운 핵산을
가지는 것.
5.1.1. Not all species of bacteria are equally efficient
at..
- 형질전환은 자연에서 쉽게 일어나지 않음.
- 실험실에서 소수의 균; Bacillus, Streptococcus
속의 일부만이 가능함.
=> 정교한 DNA의 흡착과 흡수기구를 가짐.
- 극소량의 핵산을 흡수할 수 있음.
=> 인위적 형질전환시에 박테리아 세포는
물리/화학적 처리가 요구됨.
=> competent
cell로 되어야함.
5.1.2. Preparation of competent E.coli cells
- 1970년대 초에 개발됨
=> in ice-cold salt solution
=> 50 mM CaCl2/RbCl2
are effective
=> 정확한 기능/기작은 잘모름
=> CaCl2/RbCl2가
세포의 외부에 DNA의 침착을 유도(?)
=> 세포벽의 변화를
유도 DNA결합촉진(?)
=> CaCl2/RbCl2
는 핵산의 부착을 촉진
=> NOT
UPTAKE YET!!! (그림 5. 3)
=>
DNA uptake by raising the temperature to 42C
=> 이러한 열 충격에 의한 흡수가 왜(??); not yet known fully.
5.1.3. Selection for transformed cells
- 형질전환은 그다지 효율적이지 않음.
=> uptake only 0.01% of all the
molecules
=>그러나 1 ng의
DNA로 1,000-10,000의 형질전환 박테리아를 생산가능함
=>
전체 세포로 볼때는 극히 일부에 해당함.
- 어떻게 형질과 비형질전환체를 구별하는가
=> use of a selectable marker carried
by the PLASMID
=> 그러한
마커는 형질전환체가 보여 줄 수 있는 새로운 형질이다
(one that is not possed by a non-transf.)
예: ampR/tetR in pBR322
=> amp/tet첨가 배지에서 콜로니 형성 (그림 5. 4)
=> 목적은 선별임.
=> 대부분의 plasmid는 선별 가능한
유전자를 가짐
=>항생제
저항성이란 이러한 유전자의 발현이 있는 때문임.
=>그 효소는 항생제의 독성을
제거
=> 형질전환
직후 발현되나 수 분이 지나야 저항성이 나타남.
=> 따라서
형질전환 후 곧 바로 항생제가 첨가된 배지에 뿌리지 않고, 일정시간
항생제가 첨가되지 않은 액체 배지에 두어야함.
=> 이후 복제와 저항성을 나타냄; 저항성 균주에 의한 집락의 형성
(그림 5. 5)
- self-ligation을 제한하기 위하여 phosphatase를
사용.
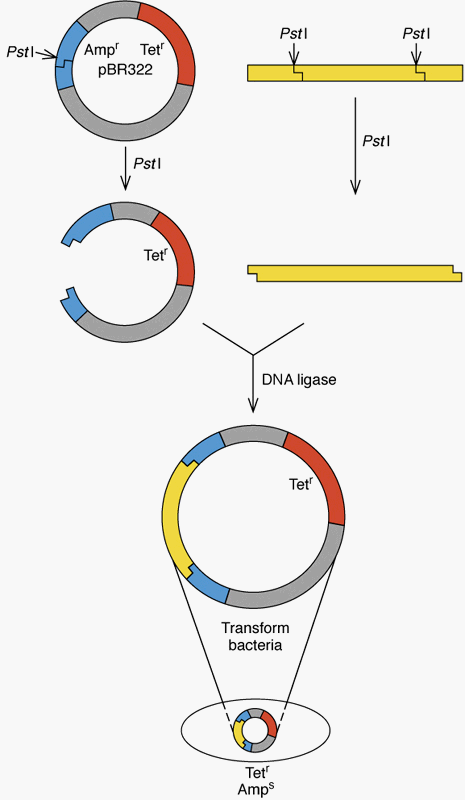
5.2. Identification of recombinants
-selectve medium; 분별가능
-self-ligated vector는 어떻게????
=> 벡터 DNA에 특정 유전자를 불활성화
시키는 법